유니스트(UNIST·울산과학기술원) 생명과학과 남덕우 교수팀은 단일세포 시퀀싱 데이터의 통합분석을 통해 폐암, 감염병 등의 질병과 관련된 유전자들을 효과적으로 선별해낼 수 있음을 확인했다고 13일 밝혔다.
단일세포 시퀀싱(scRNA-seq)은 통상적인 벌크샘플 시퀀싱(RNA-seq)과는 달리 개별 세포수준에서 유전자 발현을 분석할 수 있는 기술이다. 특히 각종 질병, 발생, 분화 등 생물학적 과정을 분석하는데 널리 활용되고 있다.
벌크샘플은 상피세포와 각종 면역세포들이 섞여있어서 세포 유형의 차이를 고려하지 못하는 단점이 있다. 반면 단일세포 시퀀싱은 세포 유형별로 순일한 유전자 발현의 변화를 측정할 수 있어 질병의 발생 기작을 보다 정확하게 분석할 수 있다. 그러나 단일세포 데이터의 높은 잡음과 결측률, 데이터 간의 측정값의 차이(배치효과)로 인해 실제로 질병유전자의 분석에 얼마나 효과적인지 확인되지 않았다.
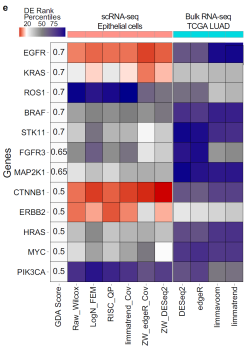
연구팀은 다양한 시뮬레이션 실험과 단일세포 데이터 분석을 통해 46가지 통합분석 방법을 비교했다. 특히, 국내에서 생산한 1기 폐암환자들의 단일세포(상피세포) 데이터를 세포유형별로 통합분석했다. 이를 통해 기존에 폐암관련 유전자로 보고된 90여 개 유전자들이 통계적으로 높은 순위를 가짐을 확인할 수 있었다. 이는 아직 밝혀지지 않은 폐암유전자들 또한 높은 순위로 선별할 수 있음을 시사한다. 이러한 결과는 기존에 진행됐던 수백 명의 벌크샘플 폐암환자의 데이터 분석에서도 달성하지 못한 결과로서 단일세포 통합분석이 폐암유전자 발굴에 매우 효과적이라는 것을 보여주고 있다.
연구팀은 이번 실험의 신빙성을 높이기 위해 대규모의 COVID-19 환자 샘플에서 단핵구 세포 10만개의 데이터를 추가로 분석했다. 통합분석으로 유전자 발현 데이터를 분석한 결과, COVID-19 바이러스의 침입에 대응하는 것으로 알려진 130여 개의 유전자가 통계적으로 높은 순위를 차지하는 것을 재차 확인할 수 있었다. 연구팀은 추가 실험을 통해 서로 다른 유형의 질병에서 단일세포 데이터의 통합분석이 질병유전자를 효과적으로 선별해 줄 수 있다는 것을 확인했다.
남덕우 생명과학과 교수는 “이번 연구 결과는 공개된 단일세포 데이터의 대규모 통합분석을 통해 새로운 질병유전자들과 작용 경로를 발굴할 수 있음을 제시해주고 있다”며 “암, 코로나19 등 다양한 질병의 기작 연구에 단일세포 데이터를 세포 수준을 넘어 유전자 수준에서 더 적극적으로 활용할 필요가 있다”라고 밝혔다.
이번 연구는 UNIST 생명과학과의 Hai Nguyen 박사와 백부경 박사가 공동 1저자로서 수행했고 중견연구자 사업과 포스트게놈 다부처유전체사업에서 지원받았다. 연구결과는 세계적인 학술지 네이쳐 커뮤니케이션즈(Nature Communications)에 3월 21일자로 게재됐다.