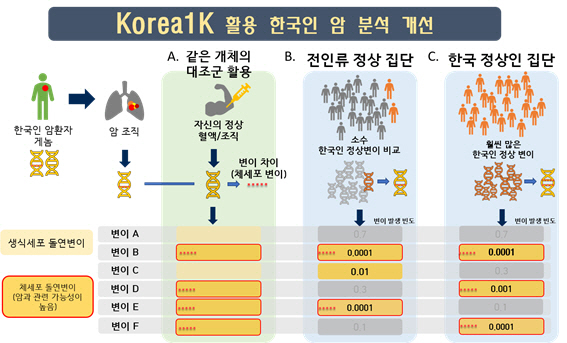
 Korea1K (한국인 1,000명 게놈 정보)를 활용한 암 분석 개선. (A) 암 환자의 암 조직 세포를 동일 환자의 정상세포를 이용해 체세포 돌연변이가 발생한 영역을 탐지. (B) 환자 정상세포 게놈 데이터가 없으면, 전 인류 정상집단과 대비해 비교(주황색 사람 픽토그램: 한국인). (C) 한국인 정상집단과 비교. 전 인류 정상집단과 대비했을 때 한국인의 암과 연관될 가능성이 높은(막대 안의 숫자가 낮을수록 연관성 높음) 체세포 돌연변이와 관련 변이(변이 B, D, E, F)를 더 잘 찾아냈다. 특히 변이 D의 경우 전 인류 정상집단에서는 암과 연관성이 낮은 변이로 판단되나 한국인 정상집단 비교 시에는 암과 연관성이 높은 변이다. /사진제공=UNIST
Korea1K (한국인 1,000명 게놈 정보)를 활용한 암 분석 개선. (A) 암 환자의 암 조직 세포를 동일 환자의 정상세포를 이용해 체세포 돌연변이가 발생한 영역을 탐지. (B) 환자 정상세포 게놈 데이터가 없으면, 전 인류 정상집단과 대비해 비교(주황색 사람 픽토그램: 한국인). (C) 한국인 정상집단과 비교. 전 인류 정상집단과 대비했을 때 한국인의 암과 연관될 가능성이 높은(막대 안의 숫자가 낮을수록 연관성 높음) 체세포 돌연변이와 관련 변이(변이 B, D, E, F)를 더 잘 찾아냈다. 특히 변이 D의 경우 전 인류 정상집단에서는 암과 연관성이 낮은 변이로 판단되나 한국인 정상집단 비교 시에는 암과 연관성이 높은 변이다. /사진제공=UNIST울산과학기술원(UNIST) 게놈산업기술센터(KOGIC)는 한국인 1,094명의 ‘전장 게놈(유전체)’과 건강검진 정보를 통합 분석한 ‘한국인 1,000명 게놈(Korea1K)’ 결과를 국제학술지 사이언스 어드밴시스 5월 27일자로 발표했다.
이 사업은 2015년 선언된 울산 만명 게놈사업의 일환으로 한국인의 모든 유전적 다양성을 지도화하기 위해 첫 번째 대규모 데이터를 공개했다. 2020년까지 1만명의 게놈 데이터를 확보할 예정이며, 모든 국민이 참여할 수 있는 일종의 국민 게놈사업이다.
이번 한국인 1,000여 명의 게놈 정보를 영국과 미국에서 2003년 완성한 인간참조표준게놈지도(표준게놈)와 비교한 결과 총 3,902만5,362개의 변이가 발견됐다. 한국인 1,000명의 게놈이 인간표준게놈과 다른 염기 약 4,000만 개를 가진다는 것이다. 특히, 이번에 발견한 변이 중 34.5%나 되는 엄청난 양의 유전자 변이가 한국인 집단 내에서 한 번만 발견되는 독특한 변이로 파악됐다.
KOGIC의 센터장인 이세민 교수는 “한국인의 개인 특이적 혹은 낮은 빈도의 희귀한 유전변이의 기능과 역할을 잘 설명하려면 더 방대한 게놈 빅데이터 확보가 절실하다”고 전했다.
한국인 1,000명 게놈(Korea1K)은 한국인의 암과 관련 있는 유전변이, 즉 ‘암 조직 특이 변이’ 예측도에서 우수한 결과를 보였다. 기존 한국인 위암 환자의 암 게놈 데이터를 한국인 1,000명 게놈(Korea1K), 다른 인족의 변이체 데이터와 비교해 암세포와 관련 있는 체세포 변이를 찾는 예측을 진행한 결과, 한국인 1,000명 게놈(Korea1K) 데이터를 활용했을 때 정확도가 가장 높았다.
이것을 분석한 최연송 연구원은 “이것은 한국인 1,000명 게놈의 실용적 가치도 매우 큼을 뜻한다”고 설명했다.
한국인 1,000명 게놈(Korea1K)에는 건강검진 결과와 유전변이 간 상관관계가 분석(전장 유전체 연관 분석, GWAS)된 결과도 담겨있다. 여기에 따르면 혈액검사로 알 수 있는 중성지방, 갑성선 호르몬 수치 등 총 11개 건강검진 항목이 15개의 게놈 영역에서 467개의 유전자 변이와 관련 있다. 이 중 4개 영역은 이번에 새롭게 발견됐으며, 9개 영역에서는 기존에 알려진 것보다 상관관계가 높은 변이를 알아냈다.
제1저자들인 생명공학과의 전성원 연구원과 박영준 연구원은 “과거의 GWAS 연구가 한정된 영역에서의 유전변이만 볼 수 있는 반면에 이 연구에서는 한국인 게놈 전체를 대량으로 읽어서 분석했기 때문에 더 정확한 유전자 연관성을 얻을 수 있었다”고 평하고 “미래엔, 대부분의 유전자 연구가 전장게놈을 가지고 행해질 것 같다”고 설명했다.
송철호 울산광역시장은 “국가 바이오 산업 발전을 위해 울산 게놈 빅데이터와 그간의 경험을 다른 국가 바이오 빅데이터 구축 사업 및 기업, 병원, 대학연구자 등에게 공유해 국내 바이오 산업 육성에 주춧돌 역할을 다할 것”이라며 “금년 내 1만 명 게놈 해독 완성을 위해 적극 지원하겠다”고 밝혔다. 울산시는 2015년부터 ‘게놈코리아 인 울산 사업’을 추진해 게놈 기반 바이오헬스산업을 육성하고 있다.
울산만명게놈사업은 참여자의 자발적 동의를 바탕으로 수집된 모든 정보를 가명화 및 익명화 절차를 통해 안전하게 관리한다. 이번 연구에서는 최소 1페타바이트(1PB=1,024테라바이트(TB))의 저장공간 (5MB 노래 파일 2억개)이 필요한 1,094명의 초대형 바이오 빅데이터를 구축했다. 한국인 1,000명 게놈 데이터는 국가적으로 공유되고 활용되기 위해 최대한 공개돼 다양한 한국인 게놈 데이터 생산에 활용될 예정이다.
한국인 게놈사업을 오랫동안 수행해온 KOGIC의 박종화 교수는 “한국인 게놈 사업은 2006년부터 과기부와 산자부의 지원으로 시작해, 국가참조표준센터·게놈연구재단·숭실대·한의학연구원·카이스트·하버드의대·케임브리지 등 다양한 국가·인족·문화 배경의 사람들이 게놈 기반 공공 빅데이터를 구축하기 위해 시작됐다”며 “과기부와 울산시의 지대한 지원에 감사드리며, 앞으로도 과학연구의 목적에 어울리게 한국 국민과 인류 전체에 활용되기를 희망한다”고 전했다.










